

图1
INTRODUCTION
•
✦
研究介绍
✦
•
研究背景
Background
“表型可塑性”是指细胞同一基因能够在不同的环境条件下表现出不同的表型或功能。其在癌症发展中常见,最近被认为是一个新兴的癌症标志。
在癌症干细胞(cancer stem cells, CSCs)的背景下,表型可塑性指的是CSCs能够在特定的微环境条件下,通过改变其表型或功能来适应不同的生存压力,例如药物治疗或转移过程中的环境。这种表型可塑性使得CSCs能够逃避治疗并促进肿瘤的复发和转移。
肿瘤细胞的可塑性归因于CSCs的存在,但目前对于CSCs是否与正常组织中的同源细胞、肿瘤的起源细胞相关联,甚至它们是否存在,都尚未达成共识。
5月31日,来自加州大学旧金山分校的Allan Balmain团队,其最新研究结果发表于《Science》期刊,研究提供了对CSCs与其正常组织对应物之间关系的更深入了解,并为癌症治疗提供了新的视角。(图1)
研究意义
Significance
对于人类肿瘤的复杂起源,它们不仅归因于暴露于环境因素,如突变原和肿瘤促进化学物质而发生,而且受到遗传和生活方式等复杂因素的影响。为了模拟这种复杂的病因,这项研究通过两个不同品系小鼠的杂交,创建了一个遗传异质性的群体,并让它们接触皮肤致癌的启动剂和促进剂。这种方法便于研究人员利用基因表达网络分析正常皮肤和肿瘤样本,进而研究干细胞在多阶段癌变过程中的作用和演变。特别地,研究聚焦于干细胞状态在肿瘤发展中的汇聚现象,以及这些状态如何与肿瘤的细胞异质性、谱系可塑性、药物抗性和治疗结果相关联。(图2)
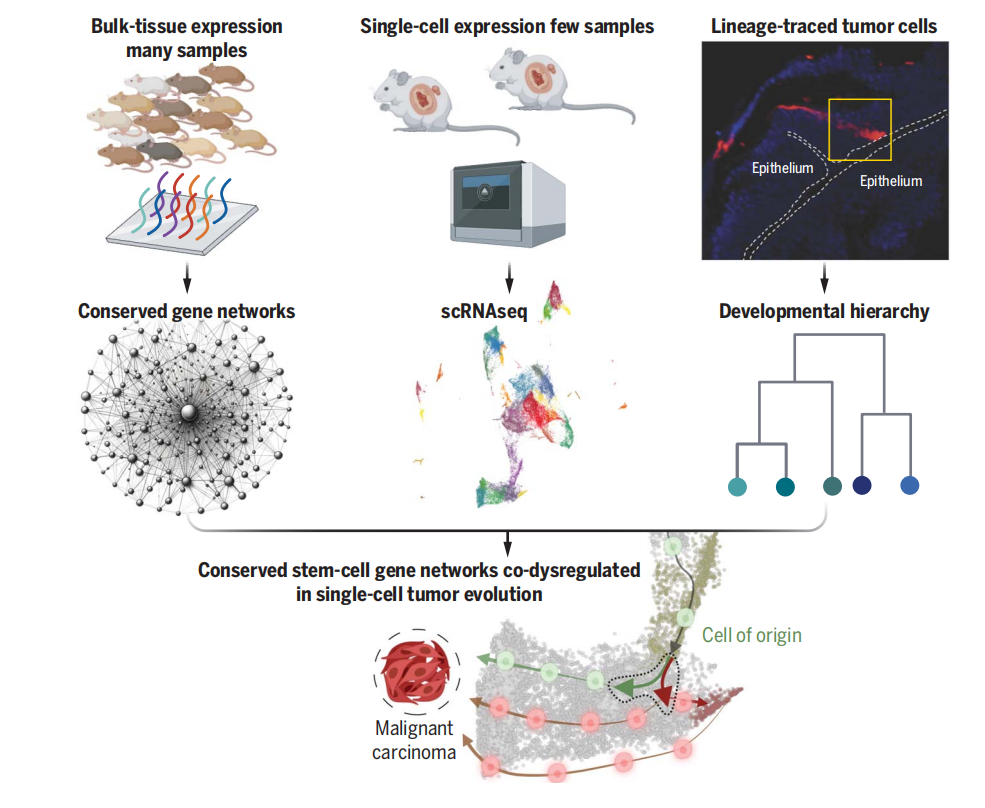
图2
METHODS
•
✦
研究方法
✦
•
1 | 基因表达网络构建:通过在小鼠中创建基因表达网络(metagenes),研究人员分析了正常皮肤和肿瘤样本中的基因表达模式。 |
2 | 单细胞RNA测序(scRNAseq):对正常皮肤、良性肿瘤和恶性肿瘤样本进行了单细胞转录组分析,以追踪干细胞状态的演变。 |
3 | 谱系追踪:使用Lgr6-eGFP-CreERT2和Rosa26LSL-Tomato报告基因系统,对Lgr6+干细胞及其后代进行长期追踪。 |
RESULTS
•
✦
研究结果
✦
•
一、
肿瘤干细胞基因表达网络的重构
研究人员利用整体组织数据采样宽度和单细胞数据高分辨率的互补性,从数百个大样本中生成一个转录组数据库,涵盖了从正常皮肤(NSk)到良性瘤前病变(皮肤乳头状瘤)到恶性肿瘤(鳞状细胞癌)以及最终转移的进展。
使用WGCNA算法对106个NSk和157个癌样本进行了无监督的基因模块分析:在肿瘤样本中,发现了一个包含1720个基因的大型模块,这个模块富含与创伤愈合相关的基因。包括几个已知的与癌变有关的干细胞标记基因,例如Sox9、Psca、Pitx1、Krt15、Krt19和Lgr6。
随后,研究人员检查了正常组织和肿瘤中这些特征明确的干细胞标记基因的相关性结构,揭示了三组基因在肿瘤中相互关联,但在匹配的正常组织中却没有。肿瘤发展过程中Lgr6元基因相关网络结构的变化揭示了与癌症进展和免疫逃逸相关的生物学功能重构。(图3)
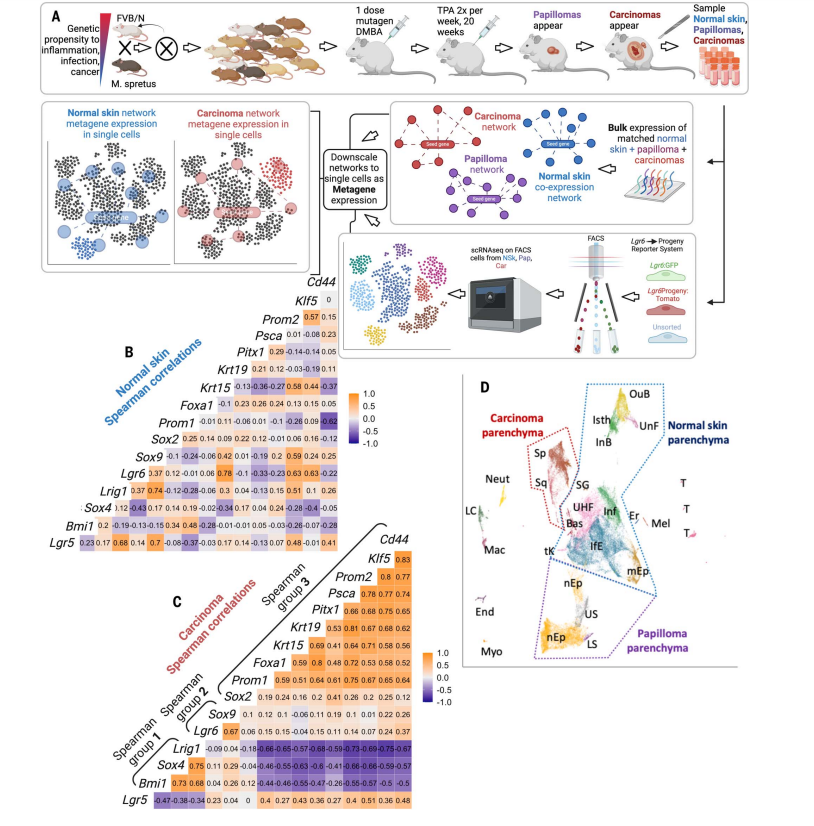
图3
作者将这些在整体组织水平上共表达的相关干细胞基因组称为“Spearman组”。这些Spearman研究小组重现了WGCNA鉴定的基因模块。重要的是,由Bmi1、Sox4和Lrig1组成的Spearman组1与Spearman组3具有很强的抗相关性,后者包含多个已知的成体干细胞标记物。
二、
多阶段癌症进展中的单细胞转录组
研究人员通过单细胞RNA测序(scRNAseq)分析了57,807个细胞,包括正常皮肤、乳头状瘤和鳞状细胞癌。他们通过无监督聚类发现了21个低分辨率的单细胞群集,其中1至4群集主要由正常皮肤、乳头状瘤和鳞状细胞癌的细胞组成。
此外,还在分析中包括了来自Lgr6-eGFP-tdTomato小鼠品系的经系谱示踪选择的NSk、良性乳头状瘤和癌症的单个上皮细胞,以标记和分离Lgr6+干细胞(eGFP+)及其直接后代(tdTomato)。
使用经典标记物对NSk细胞类型进行分类,并重新分析了仅癌症实质,结果显示了两个不同的角化细胞亚群,分别对应于鳞状(占癌症实质的20.8%)和梭形阶段(79.2%),这两个阶段均经历了上皮间质转化。
三、
基因网络可视化
研究人员探讨了如何将从大量组织样本中推断出的基因网络可视化到单个细胞中。
他们特别研究了Lgr6和Lgr5这两个与皮肤干细胞标记相关的基因,Lgr6在鳞状细胞癌中具有克隆性,而Lgr5没有。Lgr6的表达在肿瘤细胞中增加,反映了肿瘤从正常稳态向恶性发展过程中的功能转换。Lgr5在毛囊下凸起区域高表达,反映了其已知的毛囊干细胞功能,且Lgr5在癌变的所有阶段均低表达。这反映了它们在正常组织和肿瘤中的不同功能。
通过比较Lgr6和Lgr5的肿瘤特异性metagenes,实质细胞中Lgr6癌网络的metagene表达增加,而Lgr5癌网络的metagene表达减少。这表明单细胞中的metagene表达可追溯正常组织中这些干细胞标记物的功能作用,并反映恶性转化过程中发生的功能重构。
研究人员观察到,在肿瘤样本中,Spearman群组1和3的干细胞基因表达呈负相关,这表明它们可能代表两种在不同单细胞群体中表达的互斥干细胞状态。通过分析这些群组的metagenes表达,发现它们在特定的“尖峰”细胞群体中高度共定位,这些细胞群体对应于UMAP中的细胞群集33。这些“尖峰”细胞群体在表达谱系可塑性和创伤愈合相关基因方面具有高度的特异性。(图4)
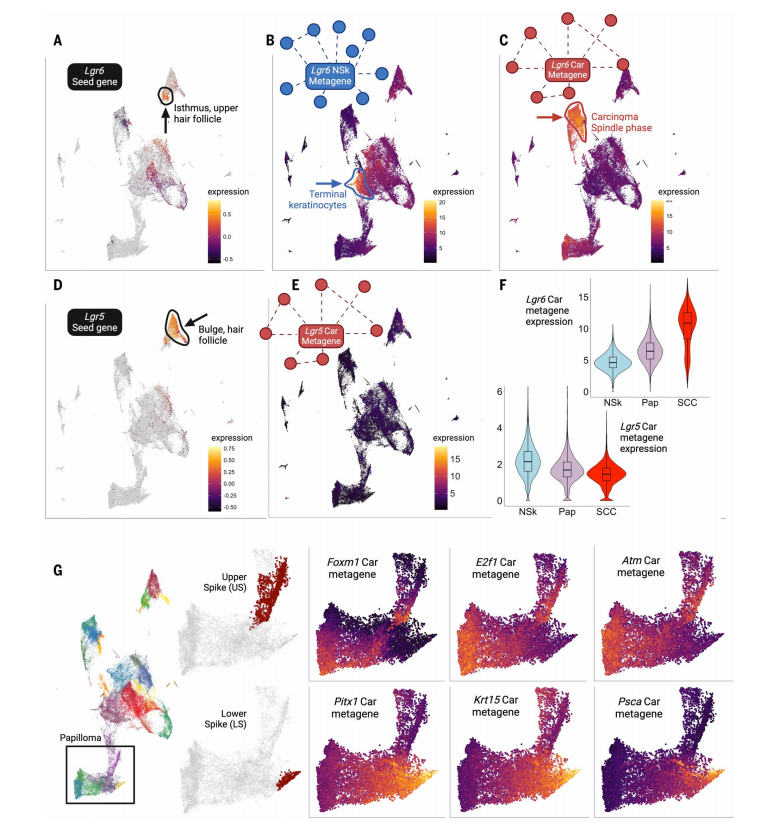
图4
四、
来源于Lgr6阳性乳头状瘤细胞的可替代干细胞群
在这一部分中,研究人员进一步探讨了Lgr6+干细胞如何与他们之前识别的两种不同的单细胞群体(上尖峰和下尖峰)相关联。通过谱系追踪和免疫荧光分析,他们发现Lgr6+干细胞在正常皮肤中重新填充了特定的区域,但在肿瘤中,这些细胞的表达更为广泛和无序。(图5)
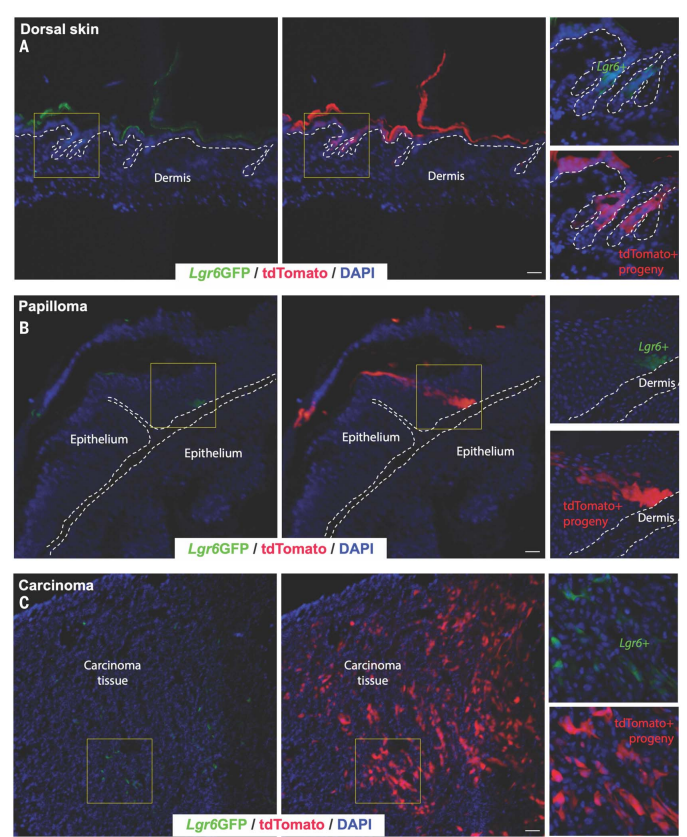
图5
研究还发现,Lgr6+干细胞产生的后代细胞在肿瘤中形成了下尖峰细胞群体,这些细胞群体高表达与谱系可塑性相关的Spearman群组3的metagenes。(图6)
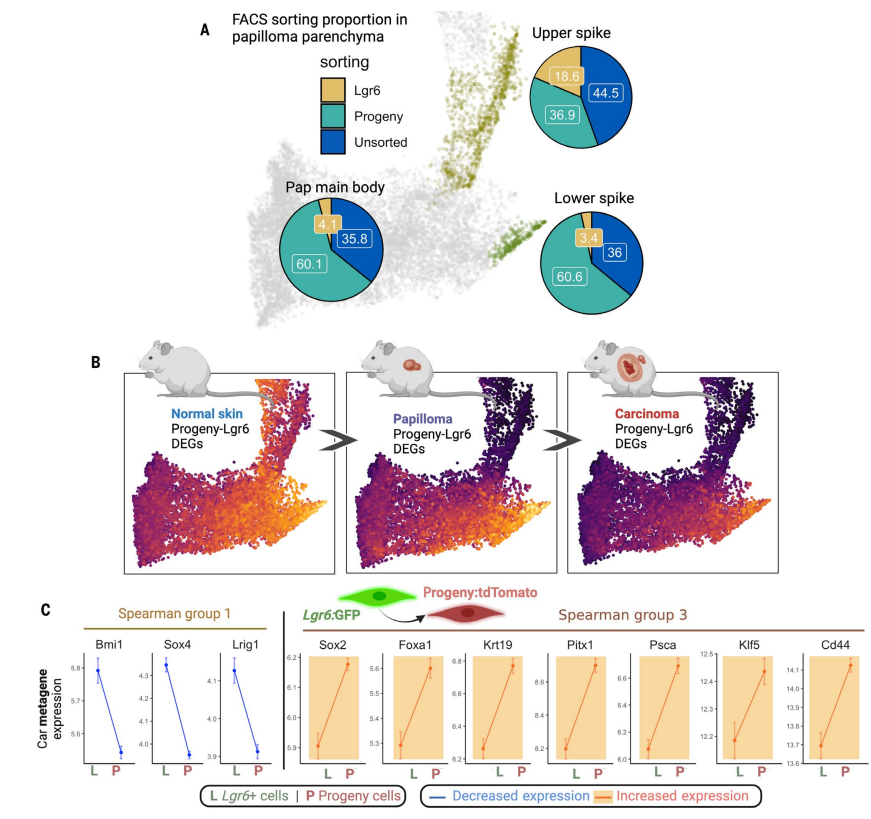
图6
为了确保单细胞metagene模式不是原始数据集中特定单细胞样本的特异和偶然结果,研究者通过scRNAseq分析了额外的独立的肿瘤样本。这些样本包含了下尖峰和上尖峰细胞,并且当与原始数据集整合时,这些额外的样本保持了上尖峰作为正常皮肤和肿瘤细胞之间的早期过渡群体的位置。
五、
下尖峰细胞状态与药物抗性之间的关联
在这一部分中,研究人员探讨了下尖峰细胞状态与药物抗性之间的关联,并在小鼠和人类肿瘤中验证了这一状态的保守性。
首先尝试确定低尖峰细胞是否会表达从药物筛选中鉴定出的经验来源的耐药标志物。检查了几种人类肿瘤类型中已知与药物抗性相关的基因,包括基底细胞癌(BCC)、前列腺腺癌(PAD)和肺腺癌。他们发现,这些肿瘤类型中与药物抗性相关的基因在小鼠的下尖峰细胞中高度表达。例如,在经过vismodegib治疗的BCC患者样本中,抗性基因Tacstd2、Ly6d和Lypd3的表达水平升高。随后使用小鼠的大量样本数据生成了这些抗性基因的肿瘤特异性metagenes,并在单细胞数据中观察到这些metagenes在下尖峰细胞中的表达。
此外,研究人员还在人类前列腺肿瘤的单细胞RNA测序数据中寻找了与下尖峰细胞状态相对应的细胞群体。他们发现,人类前列腺肿瘤中与再生能力相关的基因表达模式与小鼠下尖峰细胞状态高度一致。这表明,小鼠中发现的下尖峰细胞状态在人类肿瘤中也存在,并且可能在肿瘤发展和对治疗的响应中发挥着相似的作用。(图7)
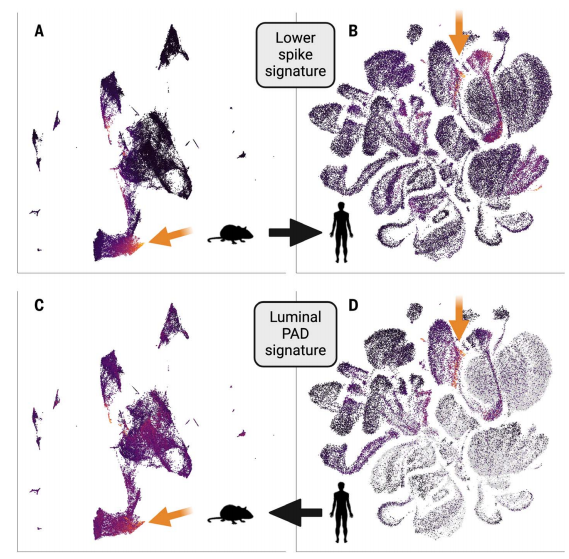
图7
研究者还探讨了化疗药物顺铂(cisplatin)对肿瘤细胞的影响。他们发现,顺铂处理可以激活下尖峰细胞状态相关的基因,这可能有助于肿瘤细胞逃避化疗药物的杀伤作用。此外,通过抑制Pp2a(一种与肿瘤抑制相关的蛋白磷酸酶)可以减少下尖峰细胞状态的表达,从而使肿瘤细胞对化疗药物更加敏感。(图8)
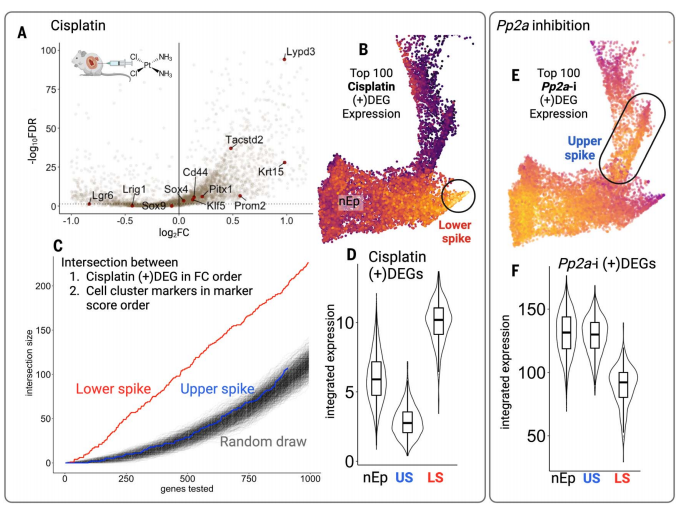
图8
这些发现揭示了下尖峰细胞状态在肿瘤发展和对治疗响应中的重要作用,为开发新的癌症治疗策略提供了潜在的靶点。通过调节这些细胞状态,可能有助于提高癌症治疗的效果,尤其是在克服肿瘤对化疗药物的抗性方面。
DISCUSSION
•
✦
研究讨论
✦
•
识别和量化肿瘤细胞的可塑性是癌症研究中的一个新兴挑战。通过在多阶段癌变过程中追踪干细胞网络的单细胞表达,研究人员识别了表达两种相反表型的不同细胞群体:一种表达快速循环的癌症特征,另一种表达慢循环的谱系可塑性癌症特征。
研究提出了一个简化的癌变模型,该模型识别了不同肿瘤进展阶段的汇聚干细胞状态。这些细胞状态背后的基因网络的调控可能为组合癌症治疗提供了潜在的靶点。
这项研究提供了对CSCs与其正常组织对应物之间关系的更深入了解,并为癌症治疗提供了新的视角。
参考文献
[1] Mark A. Taylor et al. ,Stem-cell states converge in multistage cutaneous squamous cell carcinoma development.Science384,eadi7453(2024).DOI:10.1126/science.adi7453
PROFILE
Allan Balmain
加利福尼亚大学旧金山分校Barbara Bass Bakar 癌症遗传学杰出教授
Allan Balmain教授团队多年来的主要研究目标之一是利用小鼠遗传学来了解暴露于环境致癌物质(包括化学物质和辐射)与癌症易感性之间的关系。
最近项目的重点是开发“系统遗传学”方法来分析正常组织生物学,以及癌原物暴露诱导的基因突变景观和基因表达结构的变化。这些方法旨在整合多维数据集,以提供小鼠和人类组织中正常遗传结构的网络视图,以及在良性肿瘤发展及其转移过程中发生的干扰。迄今的研究揭示了与干细胞命运决定、细胞周期控制、炎症和免疫调节相关的基因和途径的重要作用。
这些整合的系统方法代表了一种新颖且非常有前景的路径,可以识别信号通路中的关键相互作用组分,并识别与癌症相关的关键表达网络的变化。它们还将提供一个平台,利用化学诱导的具有许多点突变的肿瘤,来设计和测试新颖的联合疗法,包括放射治疗和免疫治疗,并开发响应性生物标志物。最后,分析不同致癌物质诱导的小鼠肿瘤可以更深入地了解人类癌症的病因学和突变起源。
END
文案 | 姜笑南
排版 | 姜笑南
发布|姜笑南
世界生命科学大会

RECRUIT
关注我们,获取生命科学
学界前沿|促进更多的学术交流与合作
业界前沿|促进更快的产品创新与应用
政策前沿|促进更好的治理实践与发展
